Перейти к:
МикроРНК: взгляд клинициста на состояние проблемы. Часть 1. История вопроса
https://doi.org/10.38109/2225-1685-2023-1-100-107
Аннотация
Большой объём генетической информации локализован в микроРНК, представляющих класс некодирующих РНК, образуемых из более длинных РНК-предшественников, обычно имеющих длину 19–24 нуклеотида и специфическую шпилечную структуру. Хотя исследования микроРНК были начаты относительно недавно, в настоящее время уже нет сомнений в том, что они играют важную роль в регуляции экспрессии генов на посттранскрипционном уровне в эмбриональном развитии, а также участвуют в поддержании нормальных функций клеток взрослого организма. Впервые микроРНК была обнаружена при изучении свободноживущих нематод Caenorhabditis elegans, и в свою очередь был показан новый механизм подавления экспрессии с помощью антисмысловой РНК. МикроРНК может входить в состав транскриптов, кодирующих белки, или находиться в межгенных областях генома. Изменение функциональной активности и количества микроРНК может привести к таким заболеваниям, как онкологические, сердечно-сосудистые, гинекологические, нефрологические, а также микроРНК участвует в процессе нейродегенерации и развитии психических заболеваний. Так как часть микроРНК является специфичной для определенных тканей и/или стадий развития организма, молекулы микроРНК могут рассматриваться как перспективный инструмент диагностики. Среди преимуществ данных биомаркеров можно выделить возможность обнаружения патологии в латентной стадии, малую инвазивность проводимых исследований, а также относительную устойчивость к разрушающим факторам. При этом отмечается возможность выявления микроРНК в различных биологических жидкостях: сыворотке крови, моче, семенной жидкости, слюне, грудном молоке. В настоящее время широко обсуждаются возможности применения микроРНК в таргетной терапии в связи с возможностью регулирования экспрессии генов с нежелательными свойствами либо гиперэкспрессии ингибиторов микроРНК для предотвращения негативных эффектов микроРНК, обусловливающих развитие заболевания. В первой части обзора подробно обсуждается исторический аспект изучения микроРНК, их механизм образования, особенности циркулирующих микроРНК и возможное терапевтическое влияние экзогенных микроРНК, поступающих с пищей, на организм человека.
Для цитирования:
Миронова О.Ю., Бердышева М.В., Елфимова Е.М. МикроРНК: взгляд клинициста на состояние проблемы. Часть 1. История вопроса. Евразийский Кардиологический Журнал. 2023;(1):100-107. https://doi.org/10.38109/2225-1685-2023-1-100-107
For citation:
Mironova O.I., Berdysheva M.V., Elfimova E.M. Elfimova. MicroRNA: a clinician’s view of the state of the problem. Part 1. History of the issue. Eurasian heart journal. 2023;(1):100-107. (In Russ.) https://doi.org/10.38109/2225-1685-2023-1-100-107
ВВЕДЕНИЕ
Генетическая информация реализуется в результате трёх реакций матричного синтеза: репликации, транскрипции и трансляции. Во время репликации из одной молекулы дезоксирибонуклеиновой кислоты (ДНК) образуется 2 молекулы ДНК – копии исходной ДНК, тем самым происходит сохранение и передача точной информации из поколения в поколение. Транскрипция – процесс биосинтеза всех видов рибонуклеиновой кислоты (РНК) на соответствующих участках молекул ДНК. Трансляция – завершающий процесс реализации генетической информации, при котором происходит синтез белка на рибосомах с использованием матричной РНК (мРНК) в качестве матрицы.
Сегодня ДНК считают основным носителем генетической информации, однако в последние 30 лет становится все более очевидно, что очень большой объем генетической информации также локализован в некодирующих рибонуклеиновых кислотах (РНК), наиболее изученными из которых в настоящий момент являются микроРНК [1].
МикроРНК представляют собой эндогенные небольшие (21-23 нуклеотида) рибонуклеотиды, участвующие в регуляции процесса синтеза белка из аминокислот на базе матричной РНК (мРНК), то есть трансляции, как у растений, так и у животных [2]. МикроРНК участвуют в регуляции экспрессии большинства (>60%) генов, кодирующих белки у млекопитающих, в основном за счет ее подавления. Некоторые микроРНК экспрессируются повсеместно, другие же являются специфичными для определенных тканей и/или стадий развития организма [3].
Изменения как количества, так и функциональной активности микроРНК могут приводить к развитию различных заболеваний. В частности, было показано, что микроРНК играют важную роль в развитии онкологических, сосудистых заболеваний, ангиогенезе, апоптозе [4]. Также микроРНК обладают большим потенциалом как в качестве терапевтического агента, так и в качестве собственно мишени терапевтического воздействия.
История вопроса
Caenorhabditis elegans — свободноживущая нематода (круглый червь) длиной около 1 мм. Исследования этого вида в молекулярной биологии и биологии развития начались в 1974 работами S. Brenner [5]. Широко используется как модельный организм в исследованиях по генетике, нейрофизиологии, биологии развития, вычислительной биологии. Аминокислотная и нуклеотидная последовательность генома (характерного для каждого вида организмов гаплоидного (одинарного) набора хромосом) была полностью определена и опубликована в 1998 году (дополнена в 2002).
Ambros и соавт. выяснили, что lin-4 – ген, контролирующий продолжительность личиночной стадии развития C. elegans, не кодирует никакого белка, а вместо этого продуцирует пару малых РНК [6]. Одна из РНК имела длину около 22 нуклеотидов, другая – около 61 нуклеотида. Предположительно, более длинная цепь, имеющая в своей вторичной структуре «шпильку» (структуру «стебель-петля»), являлась предшественницей более короткой (рис. 1).
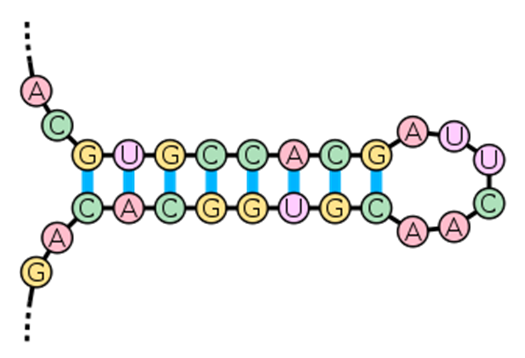
Рисунок 1. «Стебель-петля» – элемент вторичной структуры РНК
Figure 1. «Stem-loop» – an element of the secondary structure of RNA
В лабораториях Ambros и Ruvkun обнаружили, что РНК гена lin-4 имеет антисмысловую комплементарность нескольким сайтам в 3’-нетранслируемой области гена lin-14 [6][7]. Ранее считалось, что эта комплементарность в 3’-конце отвечала за репрессию гена lin-14 посредством продукции гена lin-4 [8].
Однако в лаборатории Ruvkun было обнаружено, что непосредственно микроРНК lin-4 нацелена на подавление мРНК lin-14 (рис. 2), которая содержит семь консервативных комплементарных сайтов в 3'-нетранслируемой области [6,7,9]. Удаление же этих комплементарных сайтов у мутантов с усилением функции lin-14 или мутация с потерей функции в микроРНК lin-4 нарушает нормальное уменьшение белка lin-14, которое обычно начинается в середине первой личиночной стадии (L1) [7][8][10]. Так, впервые был продемонстрирован новый механизм подавления экспрессии с помощью антисмысловой РНК.
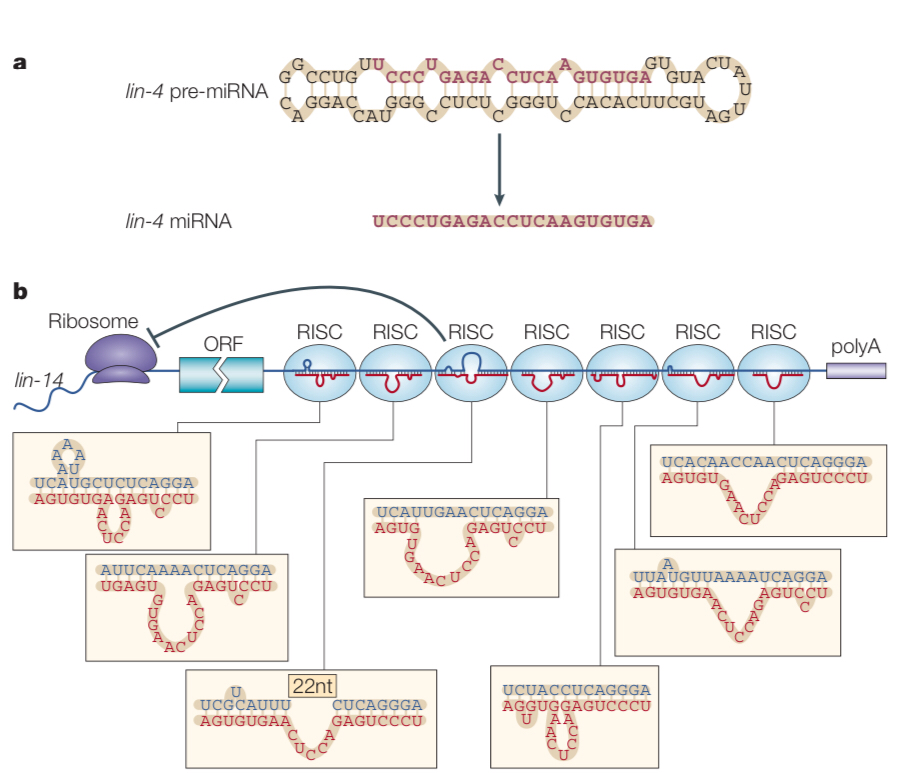
Рисунок 2. Механизм взаимодействия мРНК lin-14 и микроРНК lin-4 [11]
Синим цветом отмечена последовательность мРНК lin-14, красным отмечена последовательность микроРНК lin-4.
a – цитоплазматический процессинг, в результате которого образуется микроРНК из пре-микроРНК (данный процесс подробно изложен далее).
b – комплементарное соответствие микроРНК мРНК, вызывающее ингибирование трансляции
Figure 2. Mechanism of interaction between the lin-14 target mRNA and the lin-4 miRNA [11]
The lin-14 mRNA sequence is marked in blue, the lin-4 microRNA sequence is marked in red.
a – cytoplasmic processing, as a result of which microRNA is formed from pre-microRNA (this process is described in detail below).
b – complementary correspondence of microRNA to mRNA, causing translation inhibition
Более короткая цепь РНК lin-4 в настоящее время считается «основоположником» класса малых регулирующих РНК, называемых микроРНК [12][13][14]. В последнее время внимание многих ученых привлекает вопрос использования микроРНК как в качестве маркера различных заболеваний, так и как терапевтической мишени и агента [15-18].
В течение 7 лет после открытия РНК lin-4 геномика этого вида регуляторных РНК казалась довольно очевидной: никаких данных о наличии РНК, подобных РНК lin-4, или других подобных некодирующих РНК среди нематод получено не было. Всё изменилось после открытия let-7, другого гена C. elegans, кодирующего вторую 22-нуклеотидную регуляторную РНК. РНК let-7 отвечает за осуществление перехода C. elegans из поздней личиночной стадии во взрослую форму подобно тому, как РНК lin-4 отвечает за переход из первой личиночной стадии во вторую [19][20]. Кроме того, гомологи гена let-7 вскоре были идентифицированы в геноме человека и мухи, а РНК let-7 выделена у человека, мухи дрозофилы, а также 11 других видов двусторонне-симметричных животных [21].
Принимая во внимание схожесть ролей двуцепочечных РНК lin-4 и let-7 (относящимся к классу малых временных РНК – small temporal RNA (stRNA)) в регуляции продолжительности различных стадий развития живых организмов, вполне обоснованным было ожидать со временем открытие новых регуляторных РНК подобного типа [21]. И, в самом деле, менее чем через год три лаборатории, клонирующие малые РНК мух, червей и клеток человека, сообщили об идентификации в общей сложности более 100 дополнительных генов малых некодирующих РНК, в том числе 20 новых генов мухи дрозофилы, 30 – человека и около 60 – у червей [16-18]. РНК этих генов были похожи на малые временные РНК lin-4 и let-7 тем, что они представляли собой цепь из 22 нуклеотидов, синтезируемую на одном из плеч предшественника, имеющего в своей вторичной структуре элемент «стебель-петля». Кроме того, в большинстве своем они были эволюционно связаны между собой, одни более тесно (например, общие для C. elegans и C. briggsae), другие – менее. Необходимо отметить и важное отличие вновь идентифицированных 22-нуклеотидных РНК – они экспрессировались не на различных этапах развития, как РНК lin-4 и let-7, а в различных типах клеток. Таким образом, термин «микроРНК» использовался для обозначения малых временных РНК, а также других малых РНК с похожими свойствами, но неизвестными функциями [16-18]. Активно проводимые исследования в этом направлении позволили выявить множество дополнительных генов микроРНК млекопитающих, рыб, червей и мух [22][24-32]. Для того, чтобы упорядочить и облегчить обозначение вновь идентифицируемых генов и систематизировать данные об известных микроРНК, был создан регистр [23], на основе которого впоследствии была создана крупнейшая база данных последовательностей микроРНК miRBase (https://mirbase.org).
Номенклатура микроРНК имеет ряд правил [24], приведём в качестве примеров лишь некоторые из них. Так, префикс “miR”, за которым следует тире и число, отображает порядок наименования, причём “MiR” с заглавной буквы говорит о зрелой форме микроРНК, а с прописной – “mir” – пре-микро-РНК и при-микро-РНК [25]. Одинаковое число с добавлением буквы латинского языка отображает близость гомологов (например, miR-181a, miR-181b). Если же идентичные микроРНК закодированы в разных позициях генома, будет приписываться добавочное число через дефис (например, mir-2b-1, mir-2b-2). Принадлежность обозначается трехбуквенным префиксом (например, для человека – hsa (homo sapiens).
Большинство генов микроРНК, как и гены lin-4 и let-7 C. Elegans, относятся к участкам генома, которые находятся довольно далеко от ранее идентифицированных генов, что позволяет предположить, что они относятся к различным транскрибируемым единицам [16-18]. Кроме того, необходимо сказать, что небольшая часть (около четверти генов человеческой микроРНК) находятся в интронных участках пре-иРНК. Кроме того, синтезируемые микроРНК имеют ту же ориентацию и последовательность, что и итоговая мРНК, что позволяет предположить, что большинство этих микроРНК транскрибируются не с помощью своего собственного промотора, а подвергаются процессингу на основе интронов, что также можно видеть для многих ядрышковых РНК [28][32-36].
Другие гены микроРНК расположены в геноме и экспрессируются как мультицистронный первичный транскрипт* [12,13]. В отличие от животных, у которых гены микроРНК часто сгруппированы в кластеры и расположены внутри интронов белок-кодирующих генов, все гены микроРНК растений закодированы в межгенных спейсерах и кластеры образуют редко. При рассмотрении механизма образования и действия микроРНК необходимо подробнее остановиться на таком классе РНК, как малые интерферирующие РНК, а также на процессе РНК-интерференции в целом.
*Термин «мультицистронный транскрипт» означает молекулу мРНК, содержащую более чем одну белок-кодирующую область или цистрон. мРНК, включающая две кодирующие области, называется «бицистронным транскриптом».
Механизм образования микроРНК
Этапы общепринятого биогенеза микроРНК представлены на рисунке 3. Преобладающее большинство генов микроРНК расположены в участках более крупных кодирующих белков; остальные – в межгенных областях генома. Гены микроРНК, как правило, транскрибируются с помощью РНК-полимеразы II. К полученному протяженному первичному транскрипту (при-микроРНК) присоединяется кэп, а затем подвергается полиаденилированию [1][38][39]. РНКаза типа III Drosha разрезает при-микроРНК на 60-70 белок-связывающих пре-микроРНК, имеющих в своей структуре комплекс «стебель-петля», которые транспортируются из ядра с помощью экспортина 5 и ГТФазу Ran, относящуюся к семейству Ras-подобных ГТФаз и служащую для сопряжения транспорта с гидролизом ГТФ, что придает процессу необратимость. В цитоплазме другая РНКаза типа III, Dicer, удаляет шпильку, оставляя двойную цепь микроРНК. Она из цепей микроРНК удаляется, оставляя вторую в качестве зрелой микроРНК. Предпочтение отдается тому фрагменту, 5'-конец которого конъюгирован менее прочно. Зрелая микроРНК перемещается в комплекс сайленсинга, индуцированный РНК (RISC), где последний направляет расщепление целевой РНК с помощью Argonaute 2 (Ago2) [40]. Позиционирование осуществляется за счет комплементарности нуклеотидов 2-8, 5’-конца микроРНК, с комплементарной последовательностью целевой мРНК.
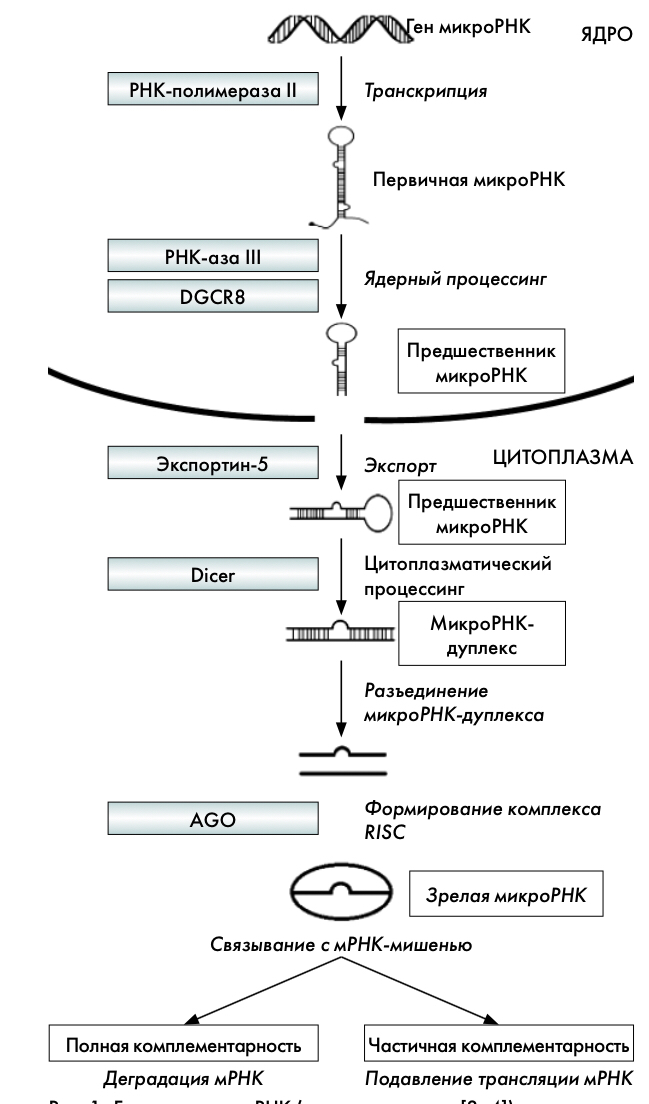
Рисунок 3. Биогенез и функция микроРНК. Механизм биосинтеза микроРНК. Описаны и иные варианты различных этапов биосинтеза микроРНК. Переработан и адаптирован [42][43]
Drosha, РНКаза типа III (эндонуклеаза); DGCR8, критическая область 8 синдрома ДиДжорджи; Dicer, другая РНКаза типа III (эндонуклеаза); RISC – комплекс сайленсинга, индуцированный РНК; Ago – Argonaute 2
Figure 3. Biogenesis and function of microRNA. Mechanism of microRNA biosynthesis. Other variants of various stages of microRNA biosynthesis are described as well. Adaptation and translation [42][43]
Drosha, RNase III (endonuclease); DiGeorge Syndrome Critical Region 8 (DGCR8); Dicer, another type RNase III (endonuclease); RISC – silencing complex induced by RNA; Ago – Argonaute 2
Эти целевые области обычно, но не всегда, расположены внутри 3’-нетранслируемой области мРНК. Важно отметить, что одна микроРНК, как правило, оказывает влияние на многие мРНК, а многие мРНК, в свою очередь, содержат участки, комплементарные нескольким микроРНК. Обычно регуляция с помощью микроРНК не носит характер «всё или ничего», а представляет собой тонкий механизм воздействия на уровень экспрессии генов. мРНК, белок и микроРНК тесно взаимосвязаны. Кроме того, есть много примеров, когда микроРНК косвенно изменяет экспрессию генов, подавляя экспрессию факторов транскрипции [41].
Важность процессинга и функционирования микроРНК широко изучается при заболеваниях различной этиологии (особенно в онкологии), в частности, в тех случаях, когда нарушается синтез и позиционирование микроРНК. Механизмы включают полиморфизмы 3’-нетранслируемой области целевой мРНК, препятствующие связыванию микроРНК, микроРНК-«ловушки», конкурентно связывающиеся со свободными местами связывания на белке, а также дефекты биогенеза микроРНК, отрицательно сказывающиеся на количестве последней [44-46].
Циркулирующие микроРНК
В работе Mitchell и соавт. было установлено, что эндогенные микроРНК находятся и циркулируют в плазме человека [47]. Авторы инкубировали образцы человеческой плазмы крови при комнатной температуре в течение 24 часов, проводили несколько циклов замораживания-оттаивания и определяли экспрессию miR-15b, miR-16 и miR-24 с помощью количественной ПЦР. В отличие от мРНК, циркулирующие микроРНК характеризовались заметно большей стабильностью [48] и устойчивостью к деградации под действием эндогенных РНКаз [49][50]. Напротив, при введении синтетических микроРНК (соответствующие микроРНК C. elegans cel-miR-39, cel-miR-54, cel-miR-238) в человеческую плазму был зафиксирован быстрый распад в течение нескольких минут. При использовании денатурирующего раствора происходила инактивация РНКаз, что, в свою очередь, позволяло защитить экзогенные микроРНК от распада. Таким образом, можно сделать вывод о том, что нативные («незащищенные») микроРНК подвержены быстрому распаду в плазме, в то время как циркулирующие микроРНК защищены и устойчивы к действию РНКаз. В этой связи был выдвинут ряд гипотез. Циркулирующие микроРНК находятся в микровезикулах (экзосомах, микрочастицах (МЧ), апоптотических тельцах), в которых они выходят в плазму крови и которые обеспечивают защиту от активности РНКаз. Другие же исследования продемонстрировали, что после изоляции микровезикул и последующего высокоскоростного центрифугирования как среды культуры клеток [51, 52], так и плазмы [53-55], микроРНК по-прежнему определялись во взвеси, лишенной микровезикул, что, в свою очередь, могло свидетельствовать о существовании невезикулярных ассоциированных комплексов микроРНК-белок/липопротеин во внеклеточных средах (рис. 4).
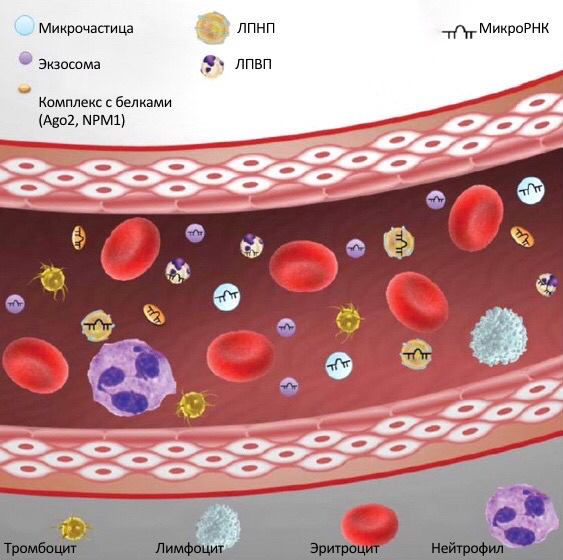
Рисунок 4. Невезикулярные ассоциированные комплексы микроРНК-белок/липопротеин во внеклеточных средах [56]
Figure 4. Extracellular miRNA that can be secreted in association with extracellular vesicles or soluble protein complexe [56]
Также циркулирующие микроРНК были выявлены и в других биологических жидкостях: в моче, семенной жидкости, слюне, грудном молоке [48][59-61].
Также в последнее время обсуждается биологический эффект экзогенных микроРНК (пища, грибы, бактерии), выявленных в кровотоке человека и попавших туда главным образом из кишечника [62].
В частности, рассматривается наличие особой роли экзогенных микроРНК в регуляции сердечно-сосудистой функции: так, при соблюдении определённой диеты наблюдается эффект кардиопротекции, который может быть результатом синергических эффектов как от эндогенных, так и экзогенных микроРНК (рис. 5) [63]. Также имеются данные о положительном терапевтическом эффекте включения в рацион питания пациентов с неалкогольной жировой болезнью печени определённых фруктов и растительных компонентов, что приводило к изменению экспрессии исследуемых микроРНК [64].
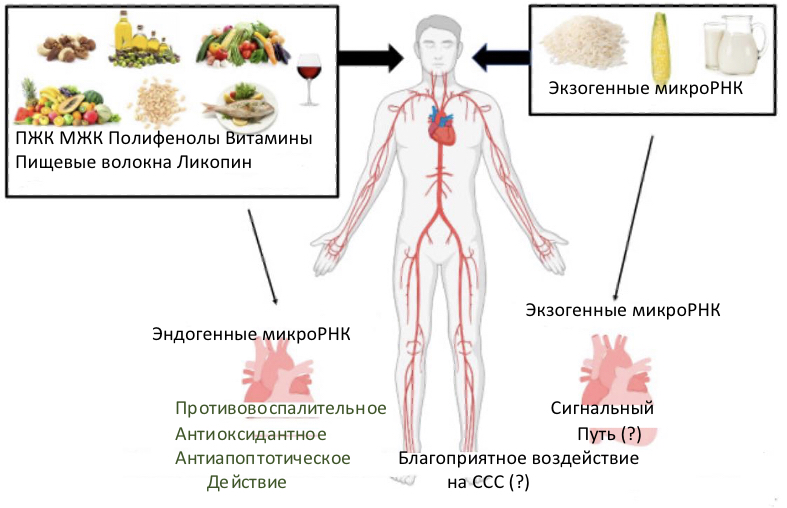
Рисунок 5. Участие микроРНК в индуцированных диетой сердечно-сосудистых преимуществах [63]
Figure 5. Participation of miRNAs in diet-induced cardiovascular benefits [63]
Заключение
Несмотря на то, что исследования микроРНК имеют довольно непродолжительную историю, уже весьма очевидна важность роли микроРНК в организме и ее влияние на большое количество процессов, протекающих в нем. Безусловно, микроРНК в ближайшие годы будет широко изучаться и дальше, ведь не остаётся сомнений в важности будущих исследований не только для более глубокого фундаментального понимания регуляции, опосредованной микроРНК, но и ее применения в диагностике и лечении различных заболеваний.
Список литературы
1. Gresele P. et al. (eds.) Antiplatelet Agents, Handbook of Experimental Pharmacology, 2012; p. 435-445.
2. Bartel DP. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 2004 Jan 23;116(2):281-97. doi: 10.1016/s0092-8674(04)00045-5. PMID: 14744438.
3. Wienholds E, Kloosterman WP, Miska E, Alvarez-Saavedra E, Berezikov E, de Bruijn E, Horvitz HR, Kauppinen S, Plasterk RH. MicroRNA expression in zebrafish embryonic development. Science. 2005 Jul 8;309(5732):310-1. doi: 10.1126/science.1114519. Epub 2005 May 26. PMID: 15919954.
4. Lai EC, Tomancak P, Williams RW, Rubin GM. Computational identification of Drosophila microRNA PMID: genes. Genome Biol. 2003;4(7):R42. doi: 10.1186/gb-2003-4-7-r42. Epub 2003 Jun 30. PMID: 12844358; PMCID: PMC193629.
5. Brenner S. The genetics of Caenorhabditis elegans. Genetics. 1974 May;77(1):71-94. doi: 10.1093/genetics/77.1.71. PMID: 4366476; PMCID: PMC1213120.
6. Lee R. C., Feinbaum R. L., Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin14. Cell 1993; 75:843-854.
7. Wightman B, Ha I, Ruvkun G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 1993 Dec 3;75(5):855-62. doi: 10.1016/0092-8674(93)90530-4. PMID: 8252622.
8. Wightman B, Bürglin TR, Gatto J, Arasu P, Ruvkun G. Negative regulatory sequences in the lin-14 3’-untranslated region are necessary to generate a temporal switch during Caenorhabditis elegans development. Genes Dev. 1991 Oct;5(10):1813-24. doi: 10.1101/gad.5.10.1813. PMID: 1916264.
9. Ha I, Wightman B, Ruvkun G. A bulged lin-4/lin-14 RNA duplex is sufficient for Caenorhabditis elegans lin-14 temporal gradient formation. Genes Dev. 1996 Dec 1;10(23):3041-50. doi: 10.1101/gad.10.23.3041. PMID: 8957004.
10. Ambros V, Horvitz HR. The lin-14 locus of Caenorhabditis elegans controls the time of expression of specific postembryonic developmental events. Genes Dev. 1987 Jun;1(4):398-414. doi: 10.1101/gad.1.4.398. PMID: 3678829.
11. He L, Hannon GJ. MicroRNAs: small RNAs with a big role in gene regulation. Nat Rev Genet. 2004 Jul;5(7):522-31. doi: 10.1038/nrg1379. Erratum in: Nat Rev Genet. 2004 Aug;5(8):631. PMID: 15211354.
12. Lagos-Quintana M, Rauhut R, Lendeckel W, Tuschl T. Identification of novel genes coding for small expressed RNAs. Science. 2001 Oct 26;294(5543):853-8. doi: 10.1126/science.1064921. PMID: 11679670.
13. Lau NC, Lim LP, Weinstein EG, Bartel DP. An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans. Science. 2001 Oct 26;294(5543):858-62. doi: 10.1126/science.1065062. PMID: 11679671.
14. Lee RC, Ambros V. An extensive class of small RNAs in Caenorhabditis elegans. Science. 2001 Oct 26;294(5543):862-4. doi: 10.1126/science.1065329. PMID: 11679672.
15. He H, Liu A, Zhang W, Yang H, Zhang M, Xu H, Liu Y, Hong B, Yan F, Yue L, Wang J, Xiao S, Xie Z, Wang T. Novel Plasma miRNAs as Biomarkers and Therapeutic Targets of Alzheimer’s Disease at the Prodromal Stage. J Alzheimers Dis. 2021;83(2):779-790. doi: 10.3233/JAD-210307. PMID: 34366343.
16. Kim TH, Lee Y, Lee YS, Gim JA, Ko E, Yim SY, Jung YK, Kang S, Kim MY, Kim H, Kim BH, Kim JH, Seo YS, Yim HJ, Yeon JE, Um SH, Byun KS. Circulating miRNA is a useful diagnostic biomarker for nonalcoholic steatohepatitis in nonalcoholic fatty liver disease. Sci Rep. 2021 Jul 19;11(1):14639. doi: 10.1038/s41598-021-94115-6. PMID: 34282172; PMCID: PMC8289842.
17. Nassar FJ, Msheik ZS, Itani MM, Helou RE, Hadla R, Kreidieh F, Bejjany R, Mukherji D, Shamseddine A, Nasr RR, Temraz SN. Circulating miRNA as Biomarkers for Colorectal Cancer Diagnosis and Liver Metastasis. Diagnostics (Basel). 2021 Feb 19;11(2):341. doi: 10.3390/diagnostics11020341. PMID: 33669508; PMCID: PMC7921943.
18. Wang J, Zhao J. MicroRNA Dysregulation in Epilepsy: From Pathogenetic Involvement to Diagnostic Biomarker and Therapeutic Agent Development. Front Mol Neurosci. 2021 Mar 12;14:650372. doi: 10.3389/fnmol.2021.650372. PMID: 33776649; PMCID: PMC7994516.
19. Reinhart BJ, Slack FJ, Basson M, Pasquinelli AE, Bettinger JC, Rougvie AE, Horvitz HR, Ruvkun G. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans. Nature. 2000 Feb 24;403(6772):901-6. doi: 10.1038/35002607. PMID: 10706289.
20. Slack FJ, Basson M, Liu Z, Ambros V, Horvitz HR, Ruvkun G. The lin41 RBCC gene acts in the C. elegans heterochronic pathway between the let-7 regulatory RNA and the LIN-29 transcription factor. Mol Cell. 2000 Apr;5(4):659-69. doi: 10.1016/s1097-2765(00)80245-2. PMID: 10882102.
21. Pasquinelli AE, Reinhart BJ, Slack F, Martindale MQ, Kuroda MI, Maller B, Hayward DC, Ball EE, Degnan B, Müller P, Spring J, Srinivasan A, Fishman M, Finnerty J, Corbo J, Levine M, Leahy P, Davidson E, Ruvkun G. Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA. Nature. 2000 Nov 2;408(6808):86- 9. doi: 10.1038/35040556. PMID: 11081512.
22. Lagos-Quintana M, Rauhut R, Yalcin A, Meyer J, Lendeckel W, Tuschl T. Identification of tissue-specific microRNAs from mouse. Curr Biol. 2002 Apr 30;12(9):735-9. doi: 10.1016/s0960-9822(02)00809-6. PMID: 12007417.
23. Griffiths-Jones S. The microRNA Registry. Nucleic Acids Res. 2004 Jan 1;32(Database issue):D109-11. doi: 10.1093/nar/gkh023. PMID: 14681370; PMCID: PMC308757.
24. Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T. A uniform system for microRNA annotation. RNA. 2003 Mar;9(3):277-9. doi: 10.1261/rna.2183803. PMID: 12592000; PMCID: PMC1370393.
25. Wright MW, Bruford EA. Naming ‘junk’: human non-protein coding RNA (ncRNA) gene nomenclature. Hum Genomics. 2011 Jan;5(2):90- 8. doi: 10.1186/1479-7364-5-2-90. PMID: 21296742; PMCID: PMC3051107
26. Mourelatos Z, Dostie J, Paushkin S, Sharma A, Charroux B, Abel L, Rappsilber J, Mann M, Dreyfuss G. miRNPs: a novel class of ribonucleoproteins containing numerous microRNAs. Genes Dev. 2002 Mar 15;16(6):720-8. doi: 10.1101/gad.974702. PMID: 11914277; PMCID: PMC155365.
27. Ambros V, Lee RC, Lavanway A, Williams PT, Jewell D. MicroRNAs and other tiny endogenous RNAs in C. elegans. Curr Biol. 2003 May 13;13(10):807-18. doi: 10.1016/s0960-9822(03)00287-2. PMID: 12747828.
28. Aravin AA, Lagos-Quintana M, Yalcin A, Zavolan M, Marks D, Snyder B, Gaasterland T, Meyer J, Tuschl T. The small RNA profile during Drosophila melanogaster development. Dev Cell. 2003 Aug;5(2):337- 50. doi: 10.1016/s1534-5807(03)00228-4. PMID: 12919683.
29. Dostie J, Mourelatos Z, Yang M, Sharma A, Dreyfuss G. Numerous microRNPs in neuronal cells containing novel microRNAs. RNA. 2003 Feb;9(2):180-6. doi: 10.1261/rna.2141503. Erratum in: RNA. 2003 May;9(5):631-2. PMID: 12554860; PMCID: PMC1370383.
30. Houbaviy HB, Murray MF, Sharp PA. Embryonic stem cell-specific MicroRNAs. Dev Cell. 2003 Aug;5(2):351-8. doi: 10.1016/s1534-5807(03)00227-2. PMID: 12919684.
31. Kim J, Krichevsky A, Grad Y, Hayes GD, Kosik KS, Church GM, Ruvkun G. Identification of many microRNAs that copurify with polyribosomes in mammalian neurons. Proc Natl Acad Sci U S A. 2004 Jan 6;101(1):360- 5. doi: 10.1073/pnas.2333854100. Epub 2003 Dec 22. PMID: 14691248; PMCID: PMC314190.
32. Lim LP, Lau NC, Weinstein EG, Abdelhakim A, Yekta S, Rhoades MW, Burge CB, Bartel DP. The microRNAs of Caenorhabditis elegans. Genes Dev. 2003 Apr 15;17(8):991-1008. doi: 10.1101/gad.1074403. Epub 2003 Apr 2. PMID: 12672692; PMCID: PMC196042.
33. Lim LP, Glasner ME, Yekta S, Burge CB, Bartel DP. Vertebrate microRNA genes. Science. 2003 Mar 7;299(5612):1540. doi: 10.1126/science.1080372. PMID: 12624257.
34. Makarova IuA, Kramerov DA. [Small nucleolar RNA genes]. Genetika. 2007 Feb;43(2):149-58. Russian. PMID: 17385312.
35. Lagos-Quintana M., Rauhut R., Meyer J., Borkhardt A., Tuschl T. New microRNAs from mouse and human. RNA 2003; 9:175-179.
36. Lai EC, Tomancak P, Williams RW, Rubin GM. Computational identification of Drosophila microRNA genes. Genome Biol. 2003;4(7):R42. doi: 10.1186/gb-2003-4-7-r42. Epub 2003 Jun 30. PMID: 12844358; PMCID: PMC193629.
37. Nana-Sinkam SP, Croce CM. MicroRNAs as therapeutic targets in cancer. Transl Res. 2011 Apr;157(4):216-25. doi: 10.1016/j.trsl.2011.01.013 Epub 2011Feb 4. PMID: 21420032
38. Cai X, Hagedorn CH, Cullen BR. Human microRNAs are processed from capped, polyadenylated transcripts that can also function as mRNAs. RNA. 2004 Dec;10(12):1957-66. doi: 10.1261/rna.7135204. Epub 2004 Nov 3. PMID: 15525708; PMCID: PMC1370684.
39. Lee Y, Kim M, Han J, Yeom KH, Lee S, Baek SH, Kim VN. MicroRNA genes are transcribed by RNA polymerase II. EMBO J. 2004 Oct 13;23(20):4051-60. doi: 10.1038/sj.emboj.7600385. Epub 2004 Sep 16. PMID: 15372072; PMCID: PMC524334.
40. Kim VN, Han J, Siomi MC. Biogenesis of small RNAs in animals. Nat Rev Mol Cell Biol. 2009 Feb;10(2):126-39. doi: 10.1038/nrm2632. PMID: 19165215.
41. Bartel DP. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 2004 Jan 23;116(2):281-97. doi: 10.1016/s0092-8674(04)00045-5. PMID: 14744438.
42. Камышова Е.С., Бобкова И.Н., Кутырина И.М. Современные представления о роли микроРНК при диабетической нефропатии: потенциальные биомаркеры и мишени таргетной терапии. Сахарный диабет. 2017;20(1):42-50. doi: 10.14341/DM8237
43. Okamura K, Hagen JW, Duan H, Tyler DM, Lai EC. The mirtron pathway generates microRNA-class regulatory RNAs in Drosophila. Cell. 2007 Jul 13;130(1):89-100. doi: 10.1016/j.cell.2007.06.028. Epub 2007 Jun 28. PMID: 17599402; PMCID: PMC2729315.
44. Sethupathy P, Collins FS. MicroRNA target site polymorphisms and human disease. Trends Genet. 2008 Oct;24(10):489-97. doi: 10.1016/j.tig.2008.07.004. Epub 2008 Sep 6. PMID: 18778868.
45. Bandiera S, Hatem E, Lyonnet S, Henrion-Caude A. microRNAs in diseases: from candidate to modifier genes. Clin Genet. 2010 Apr;77(4):306-13. doi: 10.1111/j.1399-0004.2010.01370.x. Epub 2010 Jan 5. PMID: 20132241.
46. Poliseno L, Salmena L, Zhang J, Carver B, Haveman WJ, Pandolfi PP. A coding-independent function of gene and pseudogene mRNAs regulates tumour biology. Nature. 2010 Jun 24;465(7301):1033-8. doi: 10.1038/nature09144. PMID: 20577206; PMCID: PMC3206313.
47. Mitchell P.S., Parkin R.K., Kroh E.M., Fritz B.R., Wyman S.K., Pogosova-Agadjanyan E.L. et al. Circulating microRNA as stable blood-based markers for cancer detection. Proc Natl Acad Sci USA 2008; 105:10513-10518.
48. Weber J. A., Baxter D. H., Zhang S., Huang D. Y, Huang K. H., Lee M. J. et al. The microRNA spectrum in 12 body fluids. Clin Chem 2010; 56:1733-1741.
49. Tsui NB, Ng EK, Lo YM. Stability of endogenous and added RNA in blood specimens, serum, and plasma. Clin Chem. 2002 Oct;48(10):1647-53. PMID: 12324479.
50. Wang K, Zhang S, Marzolf B, Troisch P, Brightman A, Hu Z, Hood LE, Galas DJ. Circulating microRNAs, potential biomarkers for drug-induced liver injury. Proc Natl Acad Sci U S A. 2009 Mar 17;106(11):4402-7. doi: 10.1073/pnas.0813371106. Epub 2009 Feb 25. PMID: 19246379; PMCID: PMC2657429.
51. Wang K, Zhang S, Weber J, Baxter D, Galas DJ. Export of microRNAs and microRNA-protective protein by mammalian cells. Nucleic Acids Res. 2010 Nov;38(20):7248-59. doi: 10.1093/nar/gkq601. Epub 2010 Jul 7. PMID: 20615901; PMCID: PMC2978372.
52. Turchinovich A, Weiz L, Langheinz A, Burwinkel B. Characterization of extracellular circulating microRNA. Nucleic Acids Res. 2011 Sep 1;39(16):7223-33. doi: 10.1093/nar/gkr254. Epub 2011 May 24. PMID: 21609964; PMCID: PMC3167594.
53. Zhang Y, Liu D, Chen X, Li J, Li L, Bian Z, Sun F, Lu J, Yin Y, Cai X, Sun Q, Wang K, Ba Y, Wang Q, Wang D, Yang J, Liu P, Xu T, Yan Q, Zhang J, Zen K, Zhang CY. Secreted monocytic miR-150 enhances targeted endothelial cell migration. Mol Cell. 2010 Jul 9;39(1):133-44. doi: 10.1016/j.molcel.2010.06.010. PMID: 20603081.
54. Arroyo JD, Chevillet JR, Kroh EM, Ruf IK, Pritchard CC, Gibson DF, Mitchell PS, Bennett CF, Pogosova-Agadjanyan EL, Stirewalt DL, Tait JF, Tewari M. Argonaute2 complexes carry a population of circulating microRNAs independent of vesicles in human plasma. Proc Natl Acad Sci U S A. 2011 Mar 22;108(12):5003-8. doi: 10.1073/pnas.1019055108. Epub 2011 Mar 7. PMID: 21383194; PMCID: PMC3064324.
55. Vickers KC, Palmisano BT, Shoucri BM, Shamburek RD, Remaley AT. MicroRNAs are transported in plasma and delivered to recipient cells by high-density lipoproteins. Nat Cell Biol. 2011 Apr;13(4):423-33. doi: 10.1038/ncb2210. Epub 2011 Mar 20. Erratum in: Nat Cell Biol. 2015 Jan;17(1):104. PMID: 21423178; PMCID: PMC3074610.
56. Zampetaki A, Willeit P, Drozdov I, Kiechl S, Mayr M. Profiling of circulating microRNAs: from single biomarkers to re-wired networks. Cardiovasc Res. 2012 Mar 15;93(4):555-62. doi: 10.1093/cvr/cvr266. Epub 2011 Oct 25. PMID: 22028337; PMCID: PMC3291086.
57. Arroyo JD, Chevillet JR, Kroh EM, Ruf IK, Pritchard CC, Gibson DF, Mitchell PS, Bennett CF, Pogosova-Agadjanyan EL, Stirewalt DL, Tait JF, Tewari M. Argonaute2 complexes carry a population of circulating microRNAs independent of vesicles in human plasma. Proc Natl Acad Sci U S A. 2011 Mar 22;108(12):5003-8. doi: 10.1073/pnas.1019055108. Epub 2011 Mar 7. PMID: 21383194; PMCID: PMC3064324.
58. Cortez MA, Bueso-Ramos C, Ferdin J, Lopez-Berestein G, Sood AK, Calin GA. MicroRNAs in body fluids--the mix of hormones and biomarkers. Nat Rev Clin Oncol. 2011 Jun 7;8(8):467-77. doi: 10.1038/nrclinonc.2011.76. PMID: 21647195; PMCID: PMC3423224.
59. Raposo G, Stoorvogel W. Extracellular vesicles: exosomes, microvesicles, and friends. J Cell Biol. 2013 Feb 18;200(4):373-83. doi: 10.1083/jcb.201211138. PMID: 23420871; PMCID: PMC3575529.
60. Hatmal MM, Al-Hatamleh MAI, Olaimat AN, Alshaer W, Hasan H, Albakri KA, Alkhafaji E, Issa NN, Al-Holy MA, Abderrahman SM, Abdallah AM, Mohamud R. Immunomodulatory Properties of Human Breast Milk: MicroRNA Contents and Potential Epigenetic Effects. Biomedicines. 2022 May 24;10(6):1219. doi: 10.3390/biomedicines10061219. PMID: 35740242; PMCID: PMC9219990.
61. Tomei S, Manjunath HS, Murugesan S, Al Khodor S. The Salivary miRNome: A Promising Biomarker of Disease. Microrna. 2021;10(1):29-38. doi: 10.2174/2211536610666210412154455. PMID: 33845754.
62. Chevillet JR, Lee I, Briggs HA, He Y, Wang K. Issues and prospects of microRNA-based biomarkers in blood and other body fluids. Molecules. 2014 May 14;19(5):6080-105. doi: 10.3390/molecules19056080. PMID: 24830712; PMCID: PMC6271291.
63. Collado A, Jin H, Pernow J, Zhou Z. MicroRNA: A mediator of dietinduced cardiovascular protection. Curr Opin Pharmacol. 2021 Oct;60:183-192. doi: 10.1016/j.coph.2021.07.022. Epub 2021 Aug 27. PMID: 34461563.
64. Zobeiri M, Parvizi F, Kalhori MR, Majnooni MB, Farzaei MH, Abdollahi M. Targeting miRNA by Natural Products: A Novel Therapeutic Approach for Nonalcoholic Fatty Liver. Evid Based Complement Alternat Med. 2021 Aug 13;2021:6641031. doi: 10.1155/2021/6641031. PMID: 34426744; PMCID: PMC8380168.
Об авторах
О. Ю. МироноваРоссия
Миронова Ольга Юрьевна, доктор медицинских наук, профессор кафедры факультетской терапии №1 Института клинической медицины им. Н.В. Склифосовского
ул. Большая Пироговская, д. 6, стр. 1, г. Москва 119435
М. В. Бердышева
Россия
Бердышева Мария Валерьевна, студент 5 курса
ул. Большая Пироговская, д. 6, стр. 1, г. Москва 119435
Е. М. Елфимова
Россия
Елфимова Евгения Михайловна, кандидат медицинских наук, старший научный сотрудник лаборатории апноэ сна отдела гипертонии
ул. 3-я Черепковская, д. 15 А, г. Москва 121552
Рецензия
Для цитирования:
Миронова О.Ю., Бердышева М.В., Елфимова Е.М. МикроРНК: взгляд клинициста на состояние проблемы. Часть 1. История вопроса. Евразийский Кардиологический Журнал. 2023;(1):100-107. https://doi.org/10.38109/2225-1685-2023-1-100-107
For citation:
Mironova O.I., Berdysheva M.V., Elfimova E.M. Elfimova. MicroRNA: a clinician’s view of the state of the problem. Part 1. History of the issue. Eurasian heart journal. 2023;(1):100-107. (In Russ.) https://doi.org/10.38109/2225-1685-2023-1-100-107
JATS XML































